Abstracts Publicaciones 2021
Quiénes somos
Instrucciones a autores
Responsabilidad autoría
Contacto
Portal Revistas U. de Chile
Estandarización de la técnica Real Time PCR para la detección molecular. de la mutación V617F en el gen JAK-2 en neoplasias mieloproliferativas crónicas
V617F mutation in exon 14 of Janus Kinase 2 gene (jak-2) is used as a molecular marker for the diagnosis of Philadelphia negative myeloproliferative neoplasms (Phi-) such as Polycythemia Vera (PV), Essential Thrombocythemia (ET) and Primary Myelofibrosis (MFP). To detect this mutation, we used conventional polymerase chain reaction technique (PCR), a simple and inexpensive technique, however, has some drawbacks that current technology allows to solve. During the last years, more sensitive molecular techniques have been incorporated in clinical practice to support the diagnosis, prognosis and follow-up of hematological patients. For its implementation in the clinical routine should be considered technical and economic aspects, so in this work, we evaluate the Real Time PCR technique as a diagnostic method for the detection of the Jak-2-V617F mutation, using in house primers design. Our result show that the technique implemented has a concordance index of 0.87 with the conventional PCR used in the molecular diagnosis of myeloproliferative neoplasms. In addition, it has the same specificity, greater sensitivity and, shorter execution time in relation to conventional PCR. The implementation of this diagnostic method in our Hospital is technically possible and commercially convenient.
Rev Hosp Clín Univ Chile 2019; 30: 207 - 17
Claudio Pérez N., Liliam León V., Araceli Pinto L., Gabriela Muñoz G., Jorge Alfaro L., Milton Larrondo L.
La Janus quinasa-2 (Jak-2) es parte de la familia de proteínas de transmembrana con actividad tirosina quinasa que se encuentran asociadas a receptores de citoquinas(1). Normalmente, al unirse a su ligando se produce la homo o heterodimerización del receptor y la subsecuente activación de Jak-2 mediante fosforilación. Esta activación permite el reclutamiento y la fosforilación de la proteína STAT, la que se dirige al núcleo para estimular la expresión de los genes asociados a la proliferación celular(2). En el año 2005 se reportó la mutación V617F que sustituye una guanina por una timidina en la posición 1849 en el exón 14 del gen Jak-2, traduciéndose en el reemplazo del aminoácido fenilalanina por una valina(3). La consecuencia funcional de esta mutación es una activación constitutiva de la vía de señalización Jak/STAT, generando una proliferación anormal de células hematopoyéticas y su transformación hacia neoplasias mieloproliferativas (NMP) como policitemia vera (PV), trombocitemia esencial (TE) y mielofibrosis primaria (MFP)(4). Estos trastornos clonales se encuentran estrechamente relacionados y su diagnóstico se basa en criterios aportados por la Organizaciòn Mundial de la Salud (OMS)(5). Aunque el mecanismo no es completamente comprendido, el fenotipo final de la alteración se encuentra también modulado por la alteración de otros genes, tales como MPL y CALR(6).
A partir del año 2008, la OMS incorporó la presencia de la mutación V617F en el gen de Jak-2 como criterio diagnóstico de las neoplasias mieloproliferativas, detectándose en el 95% de los pacientes con PV en 50 a 60% de los casos con TE y, en 50% de los pacientes con MFP. Adicionalmente, la mutación ha sido detectada en algunos casos raros de leucemia mielomonocítica crónica, síndrome mielodisplásico, mastocitosis sistémica y leucemia neutrofílica crónica(7), aunque no se ha establecido su valor clínico en estas patologías. Actualmente, se evalúa la cuantificación de Jak-2 V617F como una herramienta para monitorear la respuesta al tratamiento de las patologías que portan la mutación(8,9).
La determinaciòn de la mutación Jak-2 puede realizarse mediante PCR cualitativa, según la técnica descrita por Baxter en 2005(3); sin embargo, esta técnica no detecta bajas cargas mutacionales, disminuyendo su sensibilidad, y requiere del uso de bromuro de etidio, compuesto que cada vez se usa menos, considerando su potencial mutagénico y dificultades en su manipulación. En la actualidad, la mayorìas de los métodos se basan en kit comerciales de qPCR(10) por su sensibilidad y especificidad, pero son de alto costo. Por lo anterior, desarrollamos y validamos un método de PCR en tiempo real (qPCR) in house de alta sensibilidad y especificidad analítica, sustentable para nuestra institución y que valida un modelo de crecimiento en el área de la biología molecular.
MATERIALES Y MÉTODOS
1. Muestras de DNA para estudio de mutación V617F
Para la determinación de la mutación V617F en Jak- 2 mediante RT-PCR se utilizaron 46 muestras: 32 muestras de DNA almacenadas a - 80°C provenían de sangre periférica de pacientes con sospecha diagnóstica o diagnosticados como NMP y 14 muestras de DNA congelado a -80°C, provenientes de individuos sanos, donantes de Banco de Sangre.
2. PCR convencional para detección de mutación V617F
Se reanalizaron todas las muestras mediante el protocolo usado por el Laboratorio de Terapia Celular para detección cualitativa de la mutación. En el Esquema I se detallan la concentración de reactivos de amplificación (A), la secuencia de los partidores utilizados (B) y las condiciones de termociclado (C). Mediante electroforesis en gel de agarosa, se identificó la presencia de una banda de 364 pb que corresponde a la amplificación del gen Jak-2 completo y una banda de 202 pb que determina la presencia de la mutación V617F en el gen de jak-2 (Figura 1).
Esquema I. Condiciones para la reacción de PCR convencional

Figura 1. Amplificación de mutación en exón 14 del gen de Jak-2 mediante PCR convencional

3. PCR en tiempo real (qPCR) para detección de mutación V617F
La técnica de qPCR para detección y la carga mutacional V617F fue desarrollada a partir del método descrito por Larson(11) con leves modificaciones. Se utilizó el reactivo SensiMix™ II Probe kit, el que contiene todos los insumos para la reacción del qPCR, excepto los partidores y sonda. Estos últimos fueron fueron sintetizados de acuerdo a la secuencia descrita(11) y agregados exógenamente a la mezcla de reacción de qPCR. En el Esquema II se detallan la concentración de reactivos de amplificación (A), la secuencia de los partidores utilizados (B) y las condiciones de termociclado (C). La reacción de PCR se realizó en un termociclador Light Cycler™ 480II y las curvas de amplificación típicas para la mutación V617F y el alelo normal se muestran en la Figura 2. Para la cuantificación relativa de la mutación V617F, se utilizó el método de 2-··Ct descrito por Livak(24). La fórmula ·Ctjak2= Ctmut– Ctwild se utilizó para definir la carga mutacional V617F en relación al alelo Wild (no mutado). Se consideró una muestra V617F positiva o negativa cuando el Ctmut fue menor o mayor al ciclo 40 del programa de amplificación, respectivamente, asignándoles un Ctmut arbitrario de 41 a las muestras V617F negativas.
Esquema II. Condición para la reacción de PCR en tiempo real

Figura 2. Amplificación de Jak-2V617F mediante Real Time PCR
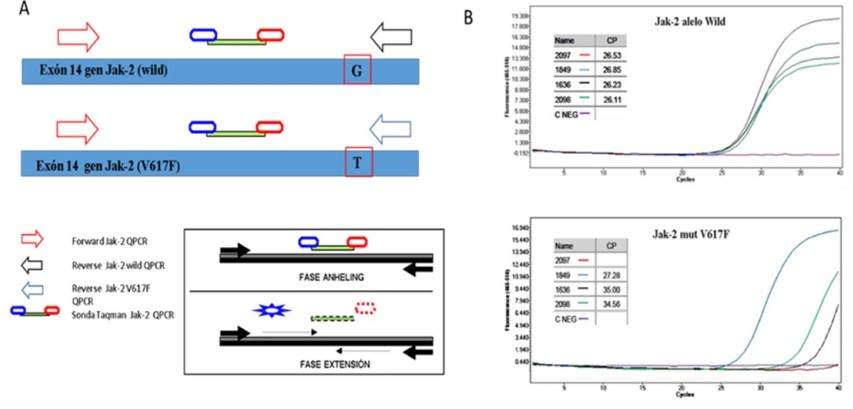
A. Estrategia de amplificación para detección de la mutación V617F. Se realizan dos reacciones: en la primera se utilizan los partidores reverse Jak-2 wild Q-PCR y forward Jak-2 Q-PCR para amplificar el alelo wild (nucleótido G en posición 1849 del gen de Jak-2) y en la segunda, se usan los partidores reverse V617F Q-PCR y forward Jak-2 Q-PCR para amplificar la mutación V617F. Ambas reacciones de PCR utilizan la sonda Taqman Jak-2 Q-PCR conjugada a flourocromo reportero FAM que permite la detección del producto amplificado mediante emisión de fluorescencia. En el recuadro, se esquematiza la hidrólisis de la sonda en la fase de extensión por acción de la DNA polimerasa que permite la detección del producto de PCR.
B. Gráfica de las curvas de amplificación de los alelos wild y V617F mostrada por el equipo Light Cycler. Las muestras 2097, 1849, 1636 y 2096 amplifican el alelo wild del gen Jak-2, mientras que sólo las muestras 1849, 1636 y 2096 amplifican el alelo V617F (muestras positivas). Además, el CP (equivalente a Ct) mostrado en recuadro, permite determinar la carga de la mutación en cada muestra mediante el método de 2-DCt.
4. Test de concordancia entre los dos métodos
Para evaluar la concordancia entre el PCR convencional y qPCR se calculó el índice Kappa (K) descrito por Cohen(12). Para la interpretación del resultado, se siguió la tabla definida por Altman(13) mostrada a continuación en la Tabla 1.
Tabla 1. Valoración del índice Kappa

RESULTADOS
1. Comparación de método de PCR convencional versus qPCR
Para validar el método de qPCR se evaluó la concordancia con el PCR convencional utilizado como método referencia. De las 46 muestras analizadas, 23 fueron positivas para ambos métodos, 20 fueron negativas y en tres hubo discrepancias, siendo 3 positivas por qPCR y negativas para PCR convencional. De acuerdo a estos valores se calculó el índice Kappa (·) que resultó en 0,87 lo que se traduce en que ambos métodos poseen un nivel o fuerza de concordancia “muy buena”. (Tabla 2)
Tabla 2. Análisis de concordancia entre PCR convencional y Real Time PCR - Cálculo de índice Kappa

2. Análisis de la especificidad y sensibilidad del método de qPCR
La mutación V617F en el gen de Jak-2 es una alteración adquirida que se asocia a NMP y está ausente en individuos sanos(14-15). En nuestro estudio, todos los individuos donantes sanos (n= 14) fueron negativos para la mutación V617F con ambos métodos, mientras que pacientes con diagnóstico confirmado de PV, MFI o TE (n=10) fueron positivos, obteniendo un 100 % de especificidad para el método. En las otras muestras Jak-2 V617 + (n=13) no se obtuvo información del diagnóstico clínico, por lo que fueron excluidas de este análisis.
En relación a la sensibilidad, se obtuvo un límite de 1/10000 (0.01%) para la detección de V617FJak-2, mediante el método de qPCR, mientras que la técnica convencional presentó un nivel de detección de 0,1% (Figura 3).
Figura 3. Sensibilidad de las técnicas moleculares utilizadas en la detección de la mutación V617F

Mediante dilución limitante, se evaluó la sensibilidad mediante PCR convencional (A) y PCR en tiempo real (B) en una muestra Jak-2V617F+.
3. Cuantificación relativa de carga mutacional V617F mediante método 2-DDCt
La carga molecular de la mutación V617F en el gen JAK2 varía considerablemente dentro de las NMP. Esta variabilidad contribuye a la determinación del fenotipo, así como al curso clínico de la patología(16). La cantidad de DNA adecuada para cuantificar la carga relativa de Jak-2V617F / Jak-2 wild por el método de 2-DDCt, estuvo en el rango de 0,1 a 100 ng. En este rango, la relación 2-DDCtse mantiene constante y no varía de acuerdo a la cantidad de DNA utilizado (Figura 4). Con estos resultados, se utilizó una cantidad de 100 ng de DNA como templado.
Figura 4. Validación de la cuantificación relativa de la mutación V617F mediante el método de 2-DDC

Los resultados de la cuantificación mostraron que las muestras positivas V617F (+) tienen un 2-··Ct medio de 0.42 con un rango de 0.0003 a 11.47, mientras que las muestras negativas tienen una media de 0.00002 con un rango de 0 a 0.00038 (Figura 5). Los puntos en gris, que en el gráfico representan las muestras discrepantes con el PCR convencional, poseen un 2-DDCt menor a 0.01, indicando una baja carga del alelo mutante (Figura 5). Dos muestras consideradas negativas que superaron el umbral, tuvieron una menor amplificación para el alelo wild (Ctwild fueron de 28.8 y 29.8), lo que incidió en el cálculo de la carga de la mutación. De acuerdo a lo anterior, la carga mutacional en una muestra debe considerar: 1) La amplificación para el alelo mutado V617F con un Ct < 40, y 2). La amplificación del alelo wild con un Ct < 28. Esto evita la confusión de valores de 2-DDCt sobre el límite considerado como Jak-2 V617F positivos.
Figura 5. Cuantificación relativa de mutación Jak-2 V617 mediante método 2-DDCt

A. Gráfico muestra los valores de 2-DDCt en muestras consideradas positivas o negativas de acuerdo a la técnica de real time PCR. El umbral de detección se marcó en 0.0003 y se muestra en línea punteada. Los puntos grises representan las tres muestras discrepantes entre el PCR convencional y el real time PCR, y los puntos blancos, las muestras negativas con un valor igual o mayor al umbral.
B. Cuadro estadístico que muestra la media, el promedio, los rangos de 2-··Ct en muestras positivas y negativas para la mutación V617F.
4. Análisis de las muestras discordantes entre los métodos
En relación a las tres muestras discordantes que fueron positivas para V617F con la técnica de PCR en tiempo real y negativas por la técnica de PCR convencional: una procedía de un paciente con diagnóstico de mielofibrosis idiopática (MFP); la segunda, de un paciente con diagnóstico de trombocitemia esencial (TE) y la tercera muestra provenía de un paciente con un hematocrito sobre 52% y hemoglobina de 18 gr/dl con criterios clínicos diagnóstico de NMP. Las discrepancias entre los resultados reflejan más bien una diferencia en la sensibilidad de ambos métodos. La amplificación de la mutación V617F (Ct) mediante qPCR en las tres muestras se detectó sobre el ciclo 34 de un 40 ciclos, una detección “tardía” que indica una baja carga mutacional y que podría explicar la negatividad en el PCR convencional.
DISCUSIÓN
Implementamos el método de qPCR in house para la detección de la mutación de V617F en el gen de Jak-2 y la comparamos con el PCR convencional utilizado en el diagnóstico molecular de NMP. Nuestros resultados muestran que existe un grado de concordancia muy bueno entre los métodos confirmando lo publicado por Larsen(11), entregando una alta sensibilidad y especificidad de la técnica de qPCR, por lo que esta metodología puede ser aplicada en el diagnóstico molecular de NMP.
Para un test molecular de uso clínico, se puede usar un kit comercial o una técnica desarrollada en el mismo laboratorio, llamado PCR in house, el cual es necesario estandarizar antes de su uso para otorgar un resultado confiable, reproducible y útil en la toma de decisiones(18). A diferencia de la forma comercial cuyo diseño generalmente permite la detección de un solo gen, los insumos utilizados en un qPCR in house pueden adaptarse para la detección de diferentes genes cambiando sólo las sondas y partidores utilizados. Además es posible utilizarlos en diferentes plataformas o instrumentos de PCR y una vez estandarizado, el método in house termina siendo más económico que un kit comercial. Una de las desventajas para el desarrollo de este tipo de procedimiento es el tiempo empleado para la estandarización y la validación del desempeño de la prueba. Existen una serie de recomendaciones para laboratorios que desarrollan técnicas moleculares y que abarcan desde las áreas y flujo de trabajo, la capacitación del personal y el equipamiento disponible hasta el uso de insumos y reactivos(19). En ese sentido, la Unidad de Biología Molecular del Servicio de Laboratorio Clínico cumple con todos los estándares para la realización de la técnica de qPCR.
Al igual que otras metodologías, la técnica de qPCR requiere de controles técnicos que garanticen la entrega de resultados confiables para el diagnóstico clínico. Como parte del procedimiento de amplificación por PCR se incluye un control positivo (DNA portador mutación V617F en el gen de Jak-2), un control negativo (DNA sin presencia de mutación V617F en el gen de Jak-2) y un control sin DNA para descartar contaminación de los reactivos. Además, como control interno se realiza un PCR para el alelo wild del gen de Jak-2, el cual debe mostrar una curva de amplificación en todas las muestras, permitiendo la validación de la calidad del DNA en la muestra problema. La ausencia de amplificación para este alelo invalida la detección de la mutación V617F y es necesario repetir la determinación o realizar una nueva extracción de DNA desde una nueva muestra de sangre periférica del paciente. Adicionalmente es recomendable que la técnica molecular implementada esté suscrita a un programa de control de la calidad externa que monitoree el desempeño del método para detección de Jak-2V617F, especialmente cuando se trabaja con técnicas in house. En comparación a la técnica de PCR convencional, el formato en tiempo real implementado presentó una mayor sensibilidad, disminuyó el tiempo de ejecución, no utilizó residuos tóxicos como el bromuro de etidio, ni generó contaminaciones post-PCR que pudiesen interferir con el resultado.
Recientemente, un panel de expertos internacionales ha propuesto la cuantificación JAK2V617F como una herramienta potencialmente útil para monitorear la respuesta al tratamiento de los nuevos fármacos dirigidos contra JAK2 en el desarrollo clínico de la NMP(6,7,18). Por otro lado, en varios estudios(16,21-23), se ha medido el impacto clínico de la cuantificación de la carga mutacional de JAK2V617F en pacientes con neoplasias mieloproliferativas crónicas Ph negativas clásicas, relacionando la carga alélica de la mutación con el desarrollo de eventos trombóticos. Dada la relevancia clínica de cuantificar la carga de la mutación V617F, empleamos el método de 2-DDCt descrito por Livak(24) para medir la carga relativa de la mutación V617F. Este método es el más apropiado para la normalización de datos y calcula la proporción de la mutación con respecto a su alelo normal sin requerir una curva de calibración con estándares de concentración conocida. Su aplicación requiere definir el rango de DNA donde la eficiencia de amplificación es la misma para ambos alelos y su relación 2-DDCt permanece constante. En nuestra técnica definimos que para la aplicación del método descrito por Livak, la cantidad ideal de DNA a utilizar se encuentra en el rango 10-100 ng. Utilizando esta estrategia de cuantificación pudimos observar que existe una gran variabilidad en la carga relativa de la mutación V617F, probablemente reflejando la heterogeneidad en las muestras estudiadas. Nuestro análisis se limita a los aspectos metodológicos; sin embargo, en trabajos futuros podría asociarse la cantidad de la mutación V617F a otros parámetros clínicos, como por ejemplo, el tipo de NMP asociada o el estado clínico del paciente. Cabe señalar que el umbral de detección fijado (0.03%) permite distinguir entre las muestras V617F positivas de las muestras negativas, pudiendo ser utilizado para evaluar la respuesta molecular al tratamiento con inhibidores de Jak-2.
Finalmente, nuestro Hospital cuenta con el equipamiento y la estructura adecuada para la aplicación rutinaria de la técnica implementada, contando con personal entrenado para el análisis e interpretación de los datos obtenidos, lo que garantiza un mejor manejo clínico, situación que puede influir positivamente en la calidad de la atención brindada a los pacientes. Además, la implementación de métodos moleculares in house crea un modelo de acción, alternativo a un kit comercial que permite incorporar nuevas técnicas para el diagnóstico de patologías con genes conocidos o la detección de alteraciones genicas descritas recientemente o de baja frecuencia para uso clínico o de investigación.
CONCLUSIÓN
Se implementó la técnica de qPCR para la detección de la mutación V617F en el gen de Jak-2, la cual puede ser empleada de forma rutinaria y confiable para el diagnóstico y seguimiento de pacientes con neoplasias mieloproliferativas crónicas.
REFERENCIAS
1. Baker SJ, Rane SG, Reddy EP. Hematopoietic cytokine receptor signaling. Oncogene 2007;26:6724.
2. Valle Mendiola A, Soto Cruz I. Vía Jak-stat: una visión general. Vertientes. Revista Especializada en Ciencias de la Salud 8(1-2).
3. Baxter EJ, Scott LM, Campbell PJ, East C, Fourouclas N, Swanton S et al. Acquired mutation of the tyrosine kinase JAK2 in human myeloproliferative disorders. Lancet 2005;365:1054-61.
4. Vainchenker W, Kralovics R. Genetic basis and molecular pathophysiology of classical myeloproliferative neoplasms. Blood 2017;129:667- 79.
5. Arber DA, Orazi A, Hasserjian R, Thiele J, Borowitz MJ, Le Beau MM et al. The 2016 revision to the World Health Organization classification of myeloid neoplasm and acute leukemia. Blood 2016;127:2391.
6. Wong WJ, Hasserjian RP, Pinkus GS, Breyfogle LJ, Mullally A, Pozdnyakova O. JAK2, CALR, MPL and ASXL1 mutational status correlates with distinct histological features in Philadelphia chromosome-negative myeloproliferative neoplasms. Haematologica 2018;103:e63-e68.
7. Tefferi A, Skoda R, Vardiman JW. Myeloproliferative neoplasms: contemporary diagnosis using histology and genetics. Nat Rev Clin Oncol 2009;6:627.
8. Barosi G, Birgegard G, Finazzi G, Griesshammer M, Harrison C, Hasselbalch HC et al. Response criteria for essential thrombocythemia and polycythemia vera: result of a European LeukemiaNet consensus conference. Blood 2009;113:4829.
9. Pardanani A, Gotlib JR, Jamieson C, Cortes JE, Talpaz M, Stone RM et al. Safety and efficacy of TG101348, a selective JAK2 inhibitor, in myelofibrosis. J Clin Oncol 2011;29:789.
10. Gong JZ, Cook JR, Greiner TC, Hedvat C, Hill CE, Lim et al. Laboratory practice guidelines for detecting and reporting JAK2 and MPL mutations in myeloproliferative neoplasms: a report of the Association for Molecular Pathology. J Molecular Diagnostics 2013;15:733-44.
11. Larsen TS, Christensen JH, Hasselbalch HC, Pallisgaard N. The JAK2 V617F mutation involves B‐and T‐lymphocyte lineages in a subgroup of patients with Philadelphia‐chromosome negative chronic myeloproliferative disorders. British J Haematology 2007;136:745-51.
12. Cohen J. A coefficient of agreement for nominal scales. Educational and psychological measurement 1960;20:37-46.
13. Altman DG. Practical statistics for medical research. CRC press: 1990.
14. Hopkins CK, Riley C, Pepkowitz S, Lopategui J R (2007). Absence of the V617F JAK2 Mutation in 181 Healthy Donors. Blood 2007;110:4664.
15. Sidon P, El Housni H, Dessars B, Heimann P. The JAK2V617F mutation is detectable at very low level in peripheral blood of healthy donors. Leukemia 2006;20:1622.
16. Fuentes AEK, Hernández-Maraver D, Lombardia L, Albendea MAC, de la Rúa AR. Impacto clínico de la cuantificación de la carga mutacional de JAK2V617F en pacientes con neoplasias mieloproliferativas crónicas Ph negativas clásicas. Medicina Clínica 2012;139:373-8.
17. Jones AV, Kreil S, Zoi K, Waghorn K, Curtis C, Zhang L et al. Widespread occurrence of the JAK2 V617F mutation in chronic myeloproliferative disorders. Blood 2005;106:2162– 8.
18. Mattocks CJ, Morris MA, Matthijs G, Swinnen E, Corveleyn A, Dequeker E et al. A standardized framework for the validation and verification of clinical molecular genetic tests. Eur J Human Genet 2010;18:1276.
19. Logan J, Logan JM, Edwards KJ, Saunders NA. (Eds.). Real-time PCR: current technology and applications. Horizon Scientific Press:2009.
20. Deininger M, Radich J, Burn TC, Huber R, Paranagama D, Verstovsek S. The effect of long-term ruxolitinib treatment on JAK2p. V617F allele burden in patients with myelofibrosis. Blood 2015;126:1551-4.
21. Borowczyk M, Wojtaszewska M, Lewandowski K, Gil L, Lewandowska M, Lehmann- Kopydłowska A et al. The JAK2 V617F mutational status and allele burden may be related with the risk of venous thromboembolic events in patients with Philadelphia-negative myeloproliferative neoplasms. Thrombosis Research 2015;135:272-80.
22. Takata Y, Seki R, Kanajii T, Nohara M, Koteda S, Kawaguchi K et al. Association between thromboembolic events and the JAK2 V617F mutation in myeloproliferative neoplasms. The Kurume Medical Journal 2011;60:89-97.
23. Carobbio A, Finazzi G, Antonioli E, Guglielmelli P, Vannucchi AM, Dellacasa et al. JAK2V617F allele burden and thrombosis: a direct comparison in essential thrombocythemia and polycythemia vera. Experimental Hematology 2009;37:1016-21.
24. Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods 2001;25:402-8.
Correspondencia:
 Dr. Claudio Pérez Núñez
Dr. Claudio Pérez Núñez
 Banco de Sangre, Hospital Clínico Universidad de Chile
Banco de Sangre, Hospital Clínico Universidad de Chile
 [email protected]
[email protected]
 56229788067
56229788067