Abstracts Publicaciones 2021
Quiénes somos
Instrucciones a autores
Responsabilidad autoría
Contacto
Portal Revistas U. de Chile
Patogenia de la infección por SARS-CoV-2
Covid-19 is an infectious disease whose etiological agent called SARS-CoV-2. It has a high contagion rate, high morbidity and mortality, and potential multi-organ compromise. Its main route of infection is by droplets, however, the infection can be acquired by other routes such as aerosols, fecal-oral and contact with fomites. Although the immune response is essential for defense against any infection, in severe Covid-19 infection, a dysregulation of the immune system occurs, which leads to marked increases of cytokines and the consequent systemic inflammatory state. Inflammation, infection, and viral shedding can compromise the lungs, blood vessels, brain, gastrointestinal (GI) tract, kidneys, heart, and liver, ultimately leading to multiple organ dysfunction and failure.
Rev Hosp Clín Univ Chile 2021; 32: 107 - 17
Nicolás Faúndes G., Karen Genskowsky V., Rocío Tordecilla F.
Los coronavirus están involucrados en enfermedades de humanos y vertebrados. El Covid-19 corresponde a una enfermedad infectocontagiosa cuyo agente etiológico ha sido denominado SARS-CoV-2 que pertenece al género Beta-coronavirus. Presentó una propagación acelerada, alcanzando proporciones epidémicas en China y posteriormente, pandémicas con amplia distribución mundial, representando actualmente un importante problema de salud pública(1-3).
En comparación a otras infecciones relacionadas, tales como SARS y MERS, posee tasas de contagio mucho más elevadas, mayor morbimortalidad y puede evolucionar a una enfermedad letal caracterizada por una respuesta inflamatoria sistémica y disfunción respiratoria(3).
ORIGEN
Se han planteado diversos reservorios y portadores intermedios desde los cuales la infección se podría haber propagado a los humanos(4-8). Los análisis de la secuencia genómica de Covid-19 han demostrado un 88% de similitud con dos coronavirus similares al síndrome respiratorio agudo severo (SARS) derivados de murciélagos, lo que indica que los mamíferos son el vínculo más probable entre Covid-19 y los humanos(6,9-11).
TRANSMISIÓN
Su principal vía de contagio es por gotitas; sin embargo, puede adquirirse la infección por otras vías tales como aerosoles, fecal-oral y contacto con fómites.
Las vías de transmisión descritas se detallan en la Tabla 1.

1. Conceptos epidemiológicos: número R0
El número de reproducción básico R0 se puede definir como el número medio de infecciones secundarias producidas por una persona. En general, se plantea que si R0 es >1, el número de casos infectados aumentará exponencialmente y provocará una epidemia o incluso una pandemia. Las estimaciones del R0 de SARS-CoV-2 oscilan entre 1,4 y 6,49, con una media de 3.28, que es más alto que el del SARS-CoV (R0 de 2 ~ 5)(4).
2. Embarazo
Diversos estudios han tenido como objetivo evaluar las características clínicas de Covid-19 en el embarazo y el potencial de transmisión vertical intrauterina de la infección. Un análisis retrospectivo de 9 pacientes embarazadas e infectadas por SARS-CoV-2 sugirió que actualmente no hay evidencia de infección intrauterina causada por transmisión vertical en mujeres que desarrollan neumonía Covid-19 al final del embarazo(12).
Otro estudio incluyó información sobre el modo de parto y el estado de infección del lactante en 655 mujeres y 666 recién nacidos. Este estudio concluyó que la infección neonatal por Covid‐19 es poco común, rara vez es sintomática y la tasa de infección no es mayor cuando el bebé nace por vía vaginal, se amamanta o permanece con la madre(13).
3. Infancia
Ha sido de gran interés comprender el papel de los niños en la transmisión del SARS-CoV-2 dadas sus implicaciones socio-económicas en relación con la reapertura de escuelas y los contactos intergeneracionales(14). Sin embargo, los niños tienen menos probabilidades de desarrollar una enfermedad grave en comparación con los adultos, aunque las razones de esto siguen sin estar claras(15).
Existe evidencia muy limitada sobre casos pediátricos que actúan como fuente de infección. Los resultados preliminares de grandes estudios de población sugieren que los niños pueden tener menos probabilidades de infectarse o infectar a otros; sin embargo, los niños muestran una mayor carga viral y una eliminación fecal más prolongada en comparación con los adultos (en algunos casos hasta más de cuatro semanas). A su vez, existe una proporción importante de niños asintomáticos, lo que genera la dificultad para poder identificar a los pacientes pediátricos infectados. Por su parte, la transmisión fecal-oral parece presentar un mayor riesgo de transmisión progresiva en los niños infectados en comparación con los adultos, dado el mayor tiempo de excreción fecal observado en varios estudios. Esto puede tener implicancias sustanciales para la propagación comunitaria en guarderías y escuelas(14,16-18).
PATOGÉNESIS
El SARS-CoV-2 es un virus de ARN pleomórfico, envuelto, de sentido positivo y monocatenario con un tamaño de genoma que varía de 26 a 32 kilobases. El tamaño de su virión es de aproximadamente 80 a 120 nm de diámetro. Posee cuatro proteínas estructurales: proteína spike (S), proteína de nucleocápside (N), proteína de “matriz” de membrana (M) y proteínas de envoltura (E)(19,20).
CICLO DE REPLICACIÓN
El ciclo de replicación de la infección por el virus del SARS-CoV-2 en la célula huésped se puede dividir en varios pasos clave: (a) Adhesión y entrada celular, (b) Transcripción de la replicasa viral, (c) Transcripción y replicación genómica, (d) Traducción de proteínas estructurales y (e) Ensamblaje y liberación de viriones(21) (ver figura 1).
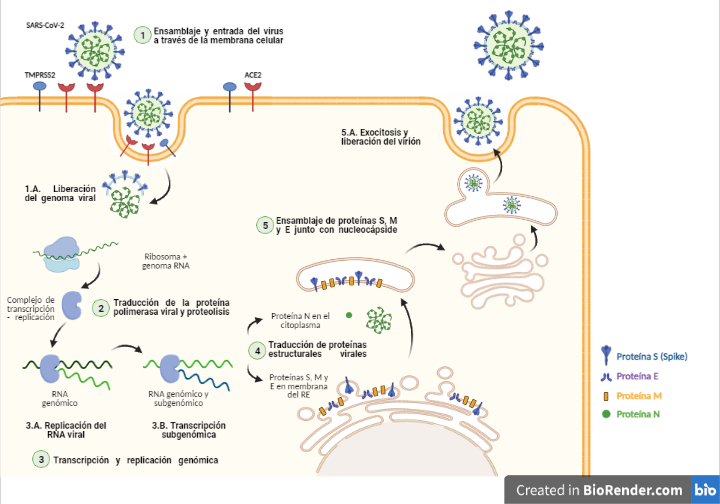
1. Adhesión y entrada:
La proteína S del SARS-CoV-2 y en particular, la región RBD, es el principal determinante del tropismo del huésped al mediar la entrada celular a través de la unión a receptores celulares. Esta interacción inicia la fusión y, por lo tanto, la infección de la célula huésped. La proteína S actúa como ligando de ACE-2 por la cual tiene una alta afinidad. Esta proteína mantiene escondido el dominio de fusión que se expone tras la acción proteolítica producida por la enzima serina 2 de proteasa transmembrana (TMPRSS-2)(21,22).
El dominio de fusión posee aminoácidos hidrófobos que permiten su inserción en la bicapa lipídica de la célula hospedadora. Esto conduce a la entrada del virión en la célula con la consiguiente entrega de la nucleocápside al citosol(23,24).
Diversos estudios han dado cuenta que la proteína S del SARS-CoV-2 con el SARS-CoV comparten un 76.5% de concordancia(1); sin embargo, existe diferencia en la composición de seis aminoácidos críticos dentro del dominio de unión de ACE-2. Esta diferencia determina que SARS-CoV-2 tenga mayor afinidad por su receptor. Por otra parte, el SARS-CoV-2 posee un sitio de escisión polibásico en la región crítica para la activación del péptido de fusión, lo que permite el clivaje por diferentes proteasas celulares adicionales (cisteínas endosómicas como la catepsina B, catepsina L, entre otras)(25). Por otra parte, se han descrito otros receptores como la lectina de tipo C CD209L (L-SIGN) y DC-SIGN que permiten el ingreso a la célula por el virus(24).
No obstante, es importante destacar que la sola presencia de ACE2 no es suficiente para hacer que las células huésped sean susceptibles a la infección. Se ha demostrado que algunas células endoteliales y líneas celulares intestinales humanas que expresan ACE2 no pudieron ser infectadas por el SARS‐CoV. Por otra parte, en algunas células sin un nivel de expresión detectable de ACE2, como los hepatocitos, también podrían infectarse por el SARS‐CoV(26,27).
2. Transcripción de la replicasa viral:
Una vez que el virus entra a la célula y se libera la nucleocápside de su envoltura, el ARN genómico (RNAg), particularmente el gen ORF1a, es transcrito y posteriormente traducido en una poliproteína denominada pp1a. A continuación, los ribosomas continúan la traducción continua en ORF1b y produce una poliproteína pp1ab más larga. La escisión proteolítica de pp1a y pp1ab genera de 15 a 16 proteínas no estructurales (NSPs) que poseen funciones específicas. Se ha descrito que la NSP3, NSP4 y NSP6 inducen el reordenamiento de la membrana celular para formar vesículas de doble membrana (DMV), donde se ensambla el complejo de transcripción-replicación del coronavirus (RTC), el cual también se encuentra constituido por otros NSPs(3,21).
En la formación de DMV y el ensamblaje de RTC, también participan factores del huésped dentro de los que se encuentra el factor GBF1 y el ARF1(21). Por último, otros genes en el tercio medio del genoma, son traducidos a RNA subgenómicos (RNAsg) que codifican proteínas estructurales tales como S, M, E y N(3,28).
3. Transcripción y replicación genómica:
El genoma de ARN de SARS-CoV-2 funciona como plantilla para que la replicasa sintetice el genoma antisentido de longitud completa (RNAsg de sentido negativo), funcionando este último como plantilla para la síntesis de nuevo ARNsg de sentido positivo(21,29).
La proteína de nucleopcápside (N) sirve como chaperona de ARN y ayuda a controlar la lectura de la plantilla de ARN. Las proteínas N de las cepas de coronavirus del SARS-CoV son fosforiladas por la glucógeno sintasa quinasa 3 (GSK3). Esta fosforilación facilita la síntesis de ARN genómico (RNAg) y RNAsg de longitud completa. Otra proteína de unión a ARN, la ribonucleoproteína nuclear heterogénea A1 (hnRNPA1), también podría unirse estrechamente a la proteína N del SARS-CoV y regular la síntesis de ARN viral(21,29).
Por otra parte, las proteínas de unión al ARN del huésped, se unen directamente a las regiones no traducidas (UTR) del genoma del coronavirus y de esta forma, regulan la replicación y la transcripción(21).
4. Traducción de proteínas estructurales:
Las proteínas S, M y E se traducen en el retículo endoplásmico (RE); sin embargo, la proteína N es traducida por los ribosomas citosólicos. Estas proteínas una vez traducidas están sujetas a modificaciones postraduccionales que finalmente las hacen completamente funcionales(20).
Tanto la proteína S como las M se modifican por glicosilación, lo que influye en la unión al receptor ACE-2, anclaje de viriones mediado por lectina y posiblemente en la neutralización por anticuerpos que los reconocen(1,24).
El plegamiento y la maduración de la proteína S ocurre en el RE modulado por chaperonas como la calnexina. El procesamiento de poliproteínas traducidas del ARN viral es esencial y lo lleva a cabo la proteasa principal (M pro, también llamada 3CL pro)(30).
5. Ensamblaje y liberación del virión:
El ensamblaje de las partículas del virión del SARS-CoV-2 tiene lugar en el compartimento intermedio ER-Golgi (ERGIC) y está mediado por la proteína de membrana (M). Las proteínas M dirigen la interacción proteína-proteína, facilitando el reclutamiento de componentes estructurales al sitio de ensamblaje. Posteriormente, las partículas de coronavirus ensambladas correctamente brotan desde el ERGIC y se transportan, utilizando vesículas. Todo esto culmina en el tráfico a través de la vía secretora y, finalmente, su liberación por exocitosis(21,31).
Existen intrincadas interacciones entre el citoesqueleto y las proteínas estructurales. Las tubulinas y el dominio citosólico de la proteína S conducen al ensamblaje y liberación exitosa de partículas virales. Además, las interacciones entre diferentes proteínas que forman parte del citoesqueleto facilitan el montaje y liberación de las partículas virales(21,31).
AFECTACIÓN DE ÓRGANOS BLANCOS
1. Pulmón
El pulmón corresponde a uno de los órganos principalmente afectados por esta infección. Una razón es que la vasta superficie del pulmón hace que sea muy susceptible a los virus inhalados, pero también se ha demostrado que el 83% de las células que expresan ACE2 son células epiteliales alveolares de tipo II (AECII), lo que sugiere su rol como reservorio para la invasión viral. Además, se comprobó que las AECII que expresan ACE2 tienen altos niveles de múltiples genes relacionados con procesos virales, incluidos genes reguladores de procesos virales, ciclo de vida viral, ensamblaje y replicación del genoma. Todo esto facilita la replicación del coronavirus en el pulmón(1).
La infección por SARS-CoV-2 en el pulmón da como resultado la acumulación de líquido dentro de los bronquiolos, alterando los recubrimientos protectores de surfactante que suelen producir los neumocitos de tipo II. Esto provoca inestabilidad alveolar y desprendimiento de células endoteliales(21).
El SDRA provoca una respuesta inmunitaria hiperactiva que localiza los neutrófilos y aumenta la liberación de citoquinas, conduciendo a la acumulación y producción de especies reactivas de oxígeno, restos celulares y proteasas. El edema es el resultado de la acumulación de proteínas en el espacio intersticial y la vasoconstricción a través de la activación plaquetaria, lo que disminuye aún más la capacidad de intercambio de oxígeno(21) (ver Figura 2).
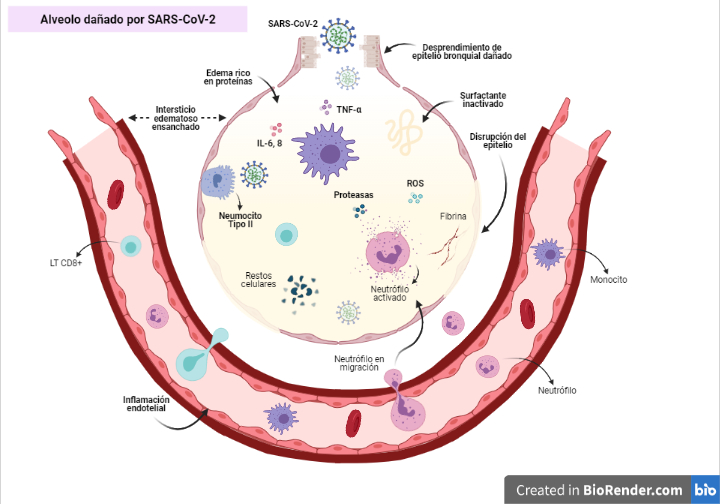
Estudios histopatológicos han demostrado la formación de membrana hialina con descamación de los neumocitos en la superficie alveolar y también infiltración intersticial de células mononucleares con la consiguiente formación de células gigantes multinucleadas(4).
2. Sistema nervioso central
El cerebro expresa ACE2, detectados en células gliales y neuronas, lo que las convierte en un objetivo potencial de Covid-19(32). Las muestras de autopsia de pacientes con Covid-19 han mostrado que el tejido cerebral secundario a la infección es hiperémico, edematoso y que la muerte neuronal estaba acompañada de partículas virales detectables(21,33,34).
Estudios previos han evidenciado que los coronavirus humanos ingresan al cerebro a partir de la circulación sistémica o mediante conexiones sinápticas y diseminación neuronal retrógrada. Esta última vía implicaría el ingreso del SARS-CoV-2 por la nariz a través del epitelio olfativo, conduciendo a la propagación transneuronal a las distintas áreas cerebrales conectadas(26).
El SARS-CoV-2 presente en la circulación sistémica puede entrar en la circulación cerebral a través de la microvasculatura. Esto facilita las interacciones de la proteína S del SARS-CoV-2 con el ACE2 presente en el endotelio capilar. La interacción de la proteína S y el ACE-2 permite que las partículas del virión pasen a través del revestimiento endotelial meníngeo para ingresar al cerebro. En el cerebro, las partículas del virión interactúan con ACE-2 que expresan las células de la neuroglia y neuronas para iniciar el ciclo de diseminación viral que causa daño neuronal(32,35).
Después de la infección por SARS-CoV-2, los linfocitos T CD4 + activados pueden secretar un factor estimulante de colonias de granulocitos y macrófagos (GM-SCF) que induce a los macrófagos a secretar citocinas proinflamatorias, perpetuando el círculo vicioso de la tormenta de citocinas tanto en la periferia como en el SNC. Esta respuesta inflamatoria da como resultado la ruptura de la barrera hematoencefálica (BHE), facilitando el ingreso de más partículas virales en el SNC. Por lo tanto, el SARS-CoV-2 puede inducir anomalías neurológicas por invasión directa del SNC o por una respuesta inflamatoria sistémica intensa indirecta que conduce a una tormenta de citoquinas(36-38).
3. Corazón
Las complicaciones cardiovasculares, como arritmias, infarto de miocardio y lesión de miocardio en pacientes con Covid-19, son los principales contribuyentes a la mortalidad(39). Varios biomarcadores se alteran en aquellos pacientes que experimentan una lesión miocárdica causada por Covid-19. La troponina plasmática y el péptido natriurético cerebral aumentan progresivamente entre los pacientes terminales con Covid-19. La troponina elevada tendría valor pronóstico para predecir arritmias malignas y requerimiento de ventilación mecánica(21).
La hipertensión y la enfermedad de las arterias coronarias son dos factores de riesgo asociados significativamente con una mayor mortalidad por Covid-19. Por su parte, el shock cardiogénico sigue siendo una complicación clave que acompaña a la insuficiencia respiratoria relacionada con la hipoxia(40).
4. Vasos sanguíneos
Recientes hallazgos sugieren el riesgo potencial de coagulopatía en pacientes que debutan con cuadros severos, más aún en aquellos que presentan comorbilidad como hipertensión, obesidad, cáncer, insuficiencia cardíaca congestiva, cáncer, etc.(41).
Se han informado varias complicaciones trombóticas que incluyen trombosis venosa profunda, accidente cerebrovascular de grandes vasos, embolia pulmonar, tromboembolismo arterial y venoso sistémico, y coagulación intravascular diseminada(41,42).
Se ha postulado que la tormenta de citoquinas y los niveles elevados de dímero D son los principales agentes asociados al desarrollo de la coagulopatía en los pacientes con Covid-19(21,41).
5. Hígado y tracto gastrointestinal
Numerosos datos apoyan la idea de que la infección por SARS-CoV-2 comúnmente afecta tanto al tracto gastrointestinal como al hígado. Es importante destacar que la ACE2 se expresa en gran medida en la superficie luminal de las células epiteliales intestinales y funciona como un correceptor para la absorción de nutrientes, en particular para la absorción de aminoácido. Por lo tanto, el intestino también podría ser un sitio de entrada importante para el SARS-CoV-2 y tener importantes implicancias para la transmisión fecal-oral y la contención de la propagación viral(1,4,21,43).
Existen reportes donde se ha podido identificar SARS-CoV-2 en las heces de pacientes con Covid- 19; sin embargo, se necesitan más estudios para aclarar la utilidad de la detección del SARS-CoV-2 en las heces y su impacto potencial en la transmisión o el manejo clínico de estos pacientes(43,44).
6. Riñón
La infección del riñón y la disfunción renal puede ocurrir con mayor frecuencia en pacientes que presentan fallas multiorgánicas. La expresión de ACE2 en los podocitos convierte al glomérulo en un objetivo directo de la infección por SARSCoV- 2, lo que lleva a la vacuolización de los podocitos, el borramiento de sus proyecciones y el desprendimiento de células. La ocupación de ACE2 por el SARS-CoV-2 podría disminuir el aclaramiento de angiotensina II, lo que contribuye aún más a la disfunción(21,44,45).
La ECA2 en las células epiteliales del túbulo contorneado proximal también las convierte en un objetivo directo de la infección por SARS-CoV-2 y las lesiones relacionadas con el virus. Este proceso podría verse agravado por una respuesta inflamatoria sistémica descontrolada (tormenta de citoquinas) e inflamación local(44-46).
CONCLUSIONES
Si bien la respuesta inmune es fundamental para la defensa frente a cualquier infección, en los casos graves de Covid-19 se liberan cantidades excesivas de citoquinas que aumentan el reclutamiento de leucocitos a múltiples órganos del cuerpo, sobre todo en los pulmones, conduciendo al síndrome de dificultad respiratoria aguda (SDRA)(21).
La infección y la diseminación viral están asociadas con enfermedades que afectan a distintos órganos diana, tales como pulmón, vasos sanguíneos, cerebro, tracto gastrointestinal (TGI), riñones, corazón e hígado. Esto último ha ampliado los criterios diagnósticos y los tratamientos para los pacientes con Covid-19(21).
Se ha planteado que tras la infección ocurre una reducción en la expresión de ACE2 y esto podría promover desregulación del sistema renina-angiotensina, inflamación y posterior tormenta de citoquinas que se presenta en muchos pacientes(25).
REFERENCIAS
1. Zhang H, Penninger JM, Li Y, Zhong N, Slutsky AS. Angiotensin ‑ converting enzyme 2 (ACE2) as a SARS‑CoV‑2 receptor: molecular mechanisms and potential therapeutic target. Intensive Care Med [Internet] 2020;46:586–90.
2. Mousavizadeh L, Ghasemi S. Genotype and phenotype of Covid-19: Their roles in pathogenesis. J Microbiol Immunol Infect [Internet] 2020 Mar 31.
3. Chen Y, Liu Q, Guo D. Emerging coronaviruses: Genome structure, replication and pathogenesis. J Med Virol 2020;92:418–23.
4. Li H, Liu S, Yu X, Tang S, Tang C. Coronavirus disease 2019 (Covid-19): current status and future perspectives. Int J Antimicrob Agents [Internet] 2020;55:105951.
5. Cui J. Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol [Internet] 2019;17:181-92.
6 Lu R, Zhao X, Li J, Niu P, Yang B, Wu H et al. Genomic characterisation and epidemiology of 2019 novel coronavirus : implications for virus origins and receptor binding. Lancet [Internet] 2020;6736:1–10.
7. Rothan HA, Byrareddy SN. The epidemiology and pathogenesis of coronavirus disease (Covid-19) outbreak. J Autoimmun [Internet] 2020;109:102433.
8. Lanka S, Emirate UA, States U. The novel Chinese coronavirus (2019-nCoV) infections: Challenges for fighting the storm. Eur J Clin Invest 2020;50:e13209.
9. Wu P, Hao X, Lau EHY, Wong JY, Leung KSM, Wu JT et al. Real-time tentative assessment of the epidemiological characteristics of novel coronavirus infections in Wuhan China, as at 22 January 2020. Euro Surveill 2020;25:2000044.
10. W Graham Carlos, Charles S Dela Cruz, Bin Cao, Susan Pasnick, Shazia Jamil. Novel Wuhan (2019-nCoV) Coronavirus. Am J Respir Crit Care Med 2020;201:P7-P8.
11. Jaimes JA, Millet JK, Stout AE, Andr NM, Whittaker GR. A tale of two viruses: the distinct spike glycoproteins of feline coronaviruses. Viruses 2020;12:83.
12. Chen H, Guo J, Wang C, Luo F, Yu X, Zhang W et al. Articles Clinical characteristics and intrauterine vertical transmission potential of Covid-19 infection in nine pregnant women: a retrospective review of medical records. J Dairy Sci [Internet] 2020;6736:1–7.
13. Id O, Grace N, Id O, Id O, Grace N, Dorling J et al. Maternal transmission of SARS-CoV-2 to the neonate, and possible routes for such transmission: A systematic review and critical analysis. BJOG 2020;127:1324-36.
14. Li X, Xu W, Dozier M, He Y. The role of children in transmission of SARS-CoV-2 : A rapid review 2020;10:1–10.
15. Cruz AT, Zeichner SL. Covid-19 in Children : Initial Characterization of the pediatric disease. Pediatrics 2020;145:e20200834.
16 Qiu H, Wu J, Hong L, Luo Y, Song Q, Chen D. Clinical and epidemiological features of 36 children with coronavirus disease 2019 (Covid-19) in Zhejiang, China: an observational cohort study. Lancet Infect Dis [Internet] 2020;2019:1–8.
17. Xing Y, Ni W, Wu Q, Li W, Li G, Wang W et al. Prolonged viral shedding in feces of pediatric patients with coronavirus disease 2019. J Microbiol Immunol Infect [Internet] 2020;53:473-80.
18. Xu Y, Li X, Zhu B, Liang H, Fang C, Gong Y et al. Characteristics of pediatric SARS-CoV-2 infection and potential evidence for persistent fecal viral shedding. Nat Med 2020;26:502-5.
19. International Committee on Taxonomy of Viruses Executive Committee, Gorbalenya AE, Krupovic M et al. The new scope of virus taxonomy: partitioning the virosphere into 15 hierarchical ranks. Nat Microbiol 2020;5:668–74.
20. Kang S, Yang M, Hong Z, Zhang L, Huang Z, Chen X et al. Crystal structure of SARSCoV- 2 nucleocapsid protein RNA binding domain reveals potential unique drug targeting sites. Acta Pharm Sin B 2020;10:1228-38.
21. Machhi J, Herskovitz J, Senan AM, Dutta D, Nath B, Oleynikov MD et al. The natural history, pathobiology and clinical manifestations of SARS-CoV-2 infections. J Neuroimmune Pharmacol 2020;21:1–28.
22. Zhang W, Du R, Li B, Zheng X, Yang X, Hu B et al. Molecular and serological investigation of 2019-nCoV infected patients : implication of multiple shedding routes. Emerg Microbes Infect 2020;9:386-9.
23. Aris JP, Guarente L, Heck DJ, Nomura M, Horiuchi T, Armstrong CM, et al. Structure of SARS Coronavirus Spike Receptor-Binding Domain Complexed with Receptor. Science 2005;309:1864-8.
24. Inhibitor P, Hoffmann M, Kleine-weber H, Schroeder S, Mu MA, Drosten C et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell 2020;181:271- 280.e8.
25. Datta PK, Liu F, Fischer T, Rappaport J, Qin X. Theranostics SARS-CoV-2 pandemic and research gaps : Understanding SARS-CoV-2 interaction with the ACE2 receptor and implications for therapy. Theranostics 2020;10:7448–64.
26. Li YC. The neuroinvasive potential of SARSCoV2 may play a role in the respiratory failure of Covid-19 patients. J Med Virol 2020;92:552-5.
27. Tay MZ, Poh CM, Rénia L, MacAry PA, Ng LFP. The trinity of Covid-19: immunity, inflammation and intervention. Nat Rev Immunol [Internet] 2020;20:363–74.
28. Li X, Geng M, Peng Y, Meng L, Lu S. Molecular immune pathogenesis and diagnosis of Covid-19. J Pharm Anal [Internet] 2020;10:102–8.
29. Zu S, Cruz JLG, Sola I, Mateos-go PA, Palacio L, Enjuanes L. Coronavirus nucleocapsid protein facilitates template switching and is required for efficient transcription 2010;84:2169–75.
30. Fung TS, Liu DX. Post-translational modifications of coronavirus proteins : roles and function. Future Virol 2018;13:405–30.
31. Siu YL, Teoh KT, Lo J, Chan CM, Kien F, Escriou N et al. The M, E, and N structural proteins of the severe acute respiratory syndrome coronavirus are required for efficient assembly, trafficking, and release of virus-like particles. J Virol 2008;82:11318– 30.
32. Baig AM, Khaleeq A, Ali U, Syeda H. Evidence of the Covid-19 virus targeting the CNS: tissue distribution, host − virus interaction, and proposed neurotropic mechanisms. ACS Chem Neurosci 2020;0–3.
33. Ling Mao, Huijuan Jin, Mengdie Wang, Yu Hu, Shengcai Chen, Quanwei He et al. Neurologic Manifestations of Hospitalized Ptients With Coronavirus Disease 2019 in Wuhan, China. JAMA Neurol 2020;77:683-90.
34. Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet 2020;395:497-506.
35. Li H, Xue Q, Xu X. Involvement of the nervous system in SARS-CoV-2 infection. Neurotox Res 2020;13:1–7.
36. Julie Helms, Stéphane Kremer, Hamid Merdji, Raphaël Clere-Jehl, Malika Schenck, Christine Kummerlen et al. Neurologic features in severe SARS-CoV-2 infection. N Engl J Med 2020;382:2268-70.
37. Moriguchi T, Harii N, Goto J, Harada D, Sugawara H, Takamino J et al. A first case of meningitis/encephalitis associated with SARS-Coronavirus-2. Int J Infect Dis 2020;94:55-8.
38. Toscano G, Palmerini F, Ravaglia S, et al. Guillain-Barré Syndrome Associated with SARS-CoV-2. N Engl J Med 2020;382:2574- 6.
39. Inciardi RM, Lupi L, Zaccone G, Italia L, Raffo M, Tomasoni D et al. Cardiac involvement in a patient with coronavirus disease 2019 (Covid-19). JAMA Cardiol 2020;5:819-24.
40. Guo T, Fan Y, Chen M, Wu X, Zhang L, He T et al. Cardiovascular implications of fatal outcomes of patients with coronavirus disease 2019 (Covid-19). JAMA Cardiol 2020;5:811- 8.
41. Barnes GD, Burnett A, Allen A, Blumenstein M, Clark NP, Cuker A et al. Thromboembolism and anticoagulant therapy during the Covid-19 pandemic: interim clinical guidance from the anticoagulation forum. J Thromb Thrombolysis 2020;50:72-81.
42. Teuwen L, Geldhof V, Pasut A, Carmeliet P. Covid-19: the vasculature unleashed. Nat Rev Immunol 2020;20:389-91.
43. Lim JK, Falck-ytter Y, El-serag HB. AGA Institute Rapid Review of the GI and Liver Manifestations of Covid-19, Meta-Analysis of International Data, and Recommendations for the Consultative Management of Patients with Covid-19. Gastroenterology 2020;159:320-334.e27.
44. Puelles VG, Lütgehetmann M, Lindenmeyer MT, Sperhake JP, Wong MN, Allweiss L et al. Multiorgan and renal tropism of SARSCoV- 2. N Engl J Med 2020;383:590-2.
45. Martinez-Rojas MA, Vega-vega O, Bobadilla XNA. Is the kidney a target of SARS-CoV-2? Am J Physiol Renal Physiol 2020;318:F1454-F1462.
46. Kursat A, Mübeccel A, Dilek A, Milena A, Brüggen WVDVM, Mahony LO et al. Immune response to SARS-CoV-2 and mechanisms of immunopathological changes in Covid-19. Allergy 2020;75:1564-81.
Correspondencia:
 Dr. Nicolás Faúndes Gandolfo
Dr. Nicolás Faúndes Gandolfo
 Sección de Inmunología, Alergias y VIH, Departamento de Medicina, HCUCH
Sección de Inmunología, Alergias y VIH, Departamento de Medicina, HCUCH
 [email protected]
[email protected]
 562 2978 8567
562 2978 8567